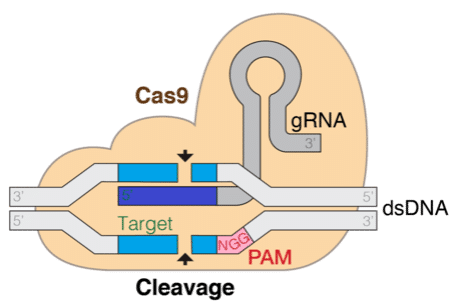
La tecnología CRISPR, basada en la edición genética, está revolucionando la comprensión y el tratamiento de enfermedades. Utilizando un mecanismo natural encontrado en bacterias, una proteína acoplada a una cadena de ARN guía (gRNA) localiza y realiza cortes específicos en el genoma objetivo. La predicción computacional de la eficiencia y especificidad del gRNA es crucial para el éxito de la edición genética.
En la reciente investigación, se adoptó un modelo de lenguaje de gran escala (LLM) preentrenado para predicción de eficiencia del gRNA. Estos modelos, conocidos por su capacidad para codificar lenguajes naturales, se están aplicando en biología computacional para resolver diversas tareas. Los LLMs, construidos sobre arquitecturas de transformadores, pueden generar secuencias que se asemejan a datos originales.
Uno de los modelos utilizados es DNABERT, un transformador preentrenado con datos de secuencias de ADN humano, que captura características del genoma permitiendo predecir eficientemente promotores, sitios de empalme y unión. Para esta investigación, se afinó DNABERT para predecir la actividad de diferentes candidatos de gRNA utilizando técnicas de Ajuste fino eficiente en parámetros (PEFT). El método LoRA (Adaptación de Baja Rango) permitió mantener los pesos del modelo preentrenado mientras introducía capas entrenables dentro de cada bloque transformador, reduciendo significativamente el número de parámetros y los requisitos de memoria de GPU.
Datos de gRNA de estudios previos fueron utilizados para entrenar el modelo en una instancia de Amazon SageMaker, empleando la biblioteca PEFT de Hugging Face. La clasificación de eficiencia fue dirigida mediante secuencias de ARN reales en cultivos celulares, buscando equilibrar la capacidad de edición del genoma sin dañar ADN no objetivo. Las métricas de evaluación incluyeron RMSE, MSE y MAE.
En las pruebas, el modelo LoRA con rango 8 mostró un desempeño cercano al modelo CRISPRon existente, un modelo de aprendizaje profundo basado en CNN. Aunque LoRA necesita mayor exploración de hiperparámetros para superar al CRISPRon, sus resultados iniciales son prometedores.
Se recomienda a los usuarios de SageMaker gestionar adecuadamente sus recursos para evitar costos innecesarios de computación no utilizada. Esta avanzada técnica en biología computacional no solo mejora la predicción de la eficiencia del gRNA de CRISPR-Cas9, sino que también abre nuevas posibilidades en aplicaciones de biología en AWS.